复旦大学彭刚组发现耳聋基因Minar2调控听觉毛细胞中胆固醇的分布与稳态
作者:CZRC 发布时间:2022/11/8 9:00:00
耳聋是最常见的感官障碍疾病[1]。目前已鉴定出120多种基因突变会引发非综合征耳聋,这反映了脊椎动物听觉系统发育和生理的复杂性。在小鼠和斑马鱼中的大规模筛选研究发现了数百个耳聋相关基因,其中部分基因的功能尚未有任何报导,提示某些关键的听觉系统发育与生理机制仍有待揭示。毛细胞作为听觉和前庭系统的机械感受器,对听觉系统至关重要[2-4]。Kiaa1024L/Minar2是一个仅在脊椎动物中保守存在的基因,它编码一个尚无任何已知功能结构域的蛋白。早前大规模筛选研究结果显示Minar2基因敲除小鼠有严重的耳聋表型[5-6],而Minar2基因在听力方面的功能研究仍属空白。
2022年11月1日,复旦大学彭刚组在eLife杂志在线发表了题为“Kiaa1024L/Minar2 is essential for hearing by regulating cholesterol distribution in hair bundles”的研究论文。该研究利用斑马鱼作为动物模型,发现胆固醇在听觉毛细胞的纤毛束中富集,并揭示了Minar2在听觉毛细胞中调节胆固醇的运输及稳态,从而保证正常的听力。值得注意的是,在此项研究论文评审期间,Bademci等在PNAS杂志报导了在多个人类非综合征耳聋家系中检测出MINAR2基因的功能缺失突变,说明MINAR2基因在人类听力发育与生理中的重要作用[7]。
在本论文前期研究中,结合已有人、小鼠和斑马鱼的数据库分析和原位杂交实验确定了minar2在斑马鱼内耳毛细胞和侧线系统毛细胞处特异性表达。接下来研究人员利用CRISPR/Cas9技术构建了minar2基因敲除的斑马鱼,并通过C-start行为学和记录听觉诱发电位实验(类似人类听力脑干反应)发现minar2-/-突变幼鱼听力异常,进一步验证了minar2在脊椎动物听力功能中发挥重要作用。由于Minar2在毛细胞处特异性表达,研究人员继而检测了毛细胞的机械敏感特性和毛细胞的基础发育。实验结果显示毛细胞的形态和功能均出现异常,包括毛细胞数量显著减少、机械电转通道异常、静纤毛细长和纤毛束异常分散(图1)。
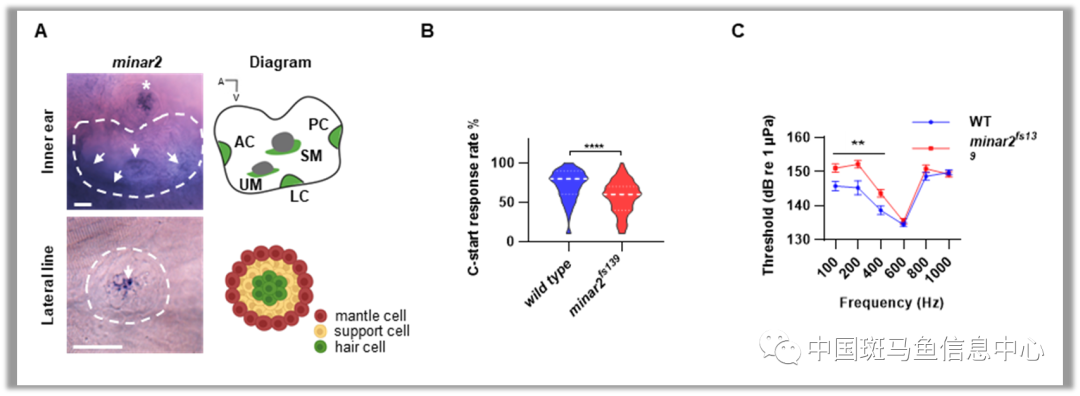
图1 minar2基因对斑马鱼的听力至关重要

图1 minar2基因对斑马鱼的听力至关重要
研究人员接下来利用Tol2转座酶系统和毛细胞特异myo6b启动子,构建毛细胞特异性表达的转基因斑马鱼。首先利用Tg(myo6b:GFP-Minar2)和Tg(myo6b:GFP-P2A-FLAG-Minar2)转基因鱼,观察到Minar2蛋白在毛细胞的顶端区域和静纤毛上定位(图2)。文献中电镜及免疫组化结果显示毛细胞顶端区域富集细胞内膜系统,其中也包括溶酶体[8-10]。利用myo6b:Lamp1-GFP特异性标记斑马鱼毛细胞中的溶酶体,实验结果显示Minar2与Lamp1的荧光信号在毛细胞顶端部分重叠,且在minar2-/-斑马鱼中溶酶体形态异常增大、变圆。这些结果均提示minar2基因的缺失导致毛细胞中溶酶体异常。
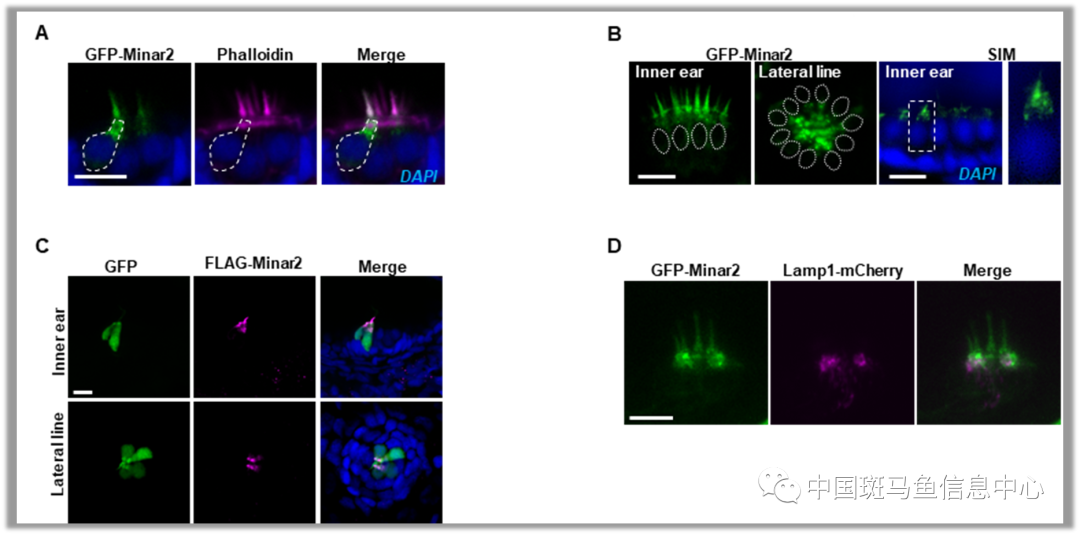
图2 Minar2的蛋白定位

图2 Minar2的蛋白定位
另一方面,研究通过生物信息学的方式检索有关Minar2分子功能的线索,发现Minar2蛋白上可能存在一个与caveolin scaffolding domain (CSD)结构域类似的序列,此CSD结构域可与胆固醇分子直接相互作用。这为深入探究Minar2在毛细胞中的机制提供了线索。产气荚膜梭状芽孢杆菌(Perfringolysin O,PFO)毒素可以结合胆固醇,D4H是一种对重组后的PFO毒素第四个结构域进行改进的生物探针,改进后可更敏感地识别胆固醇[11-15]。研究利用胆固醇探针D4H,构建了毛细胞特异Tg(myo6b:D4H-mCherry)转基因动物,首次在动物模型中实现对体内毛细胞胆固醇的标记(图3)。非常显著的是,D4H探针标记的胆固醇信号在毛细胞静纤毛和动纤毛上富集,而在毛细胞基底膜和胞体中几乎不分布。
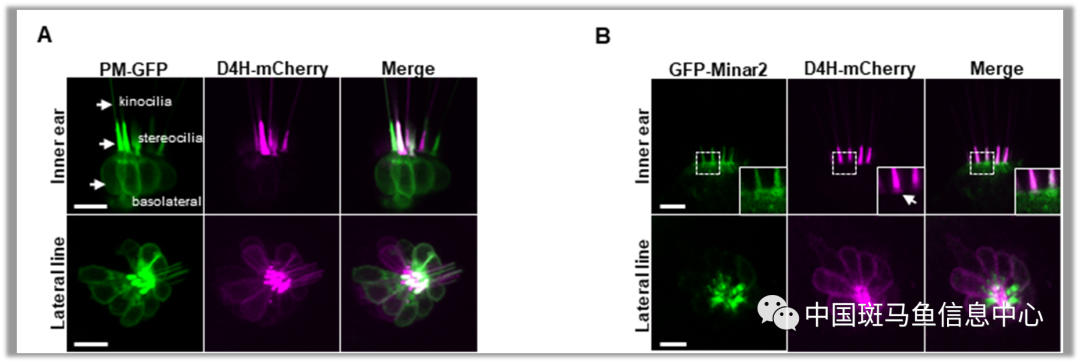
图3 毛细胞中胆固醇和Minar2的共定位

图3 毛细胞中胆固醇和Minar2的共定位
由于Minar2蛋白和胆固醇探针D4H均分布在静纤毛上,提示Minar2与胆固醇密切相关。研究进一步发现在minar2-/-斑马鱼中发现胆固醇水平显著降低,并且使用特异性药物降低胆固醇会加重minar2-/-斑马鱼毛细胞的异常;反之使用胆固醇复合物和药物干预补加胆固醇可以拯救minar2-/-斑马鱼中毛细胞以及听力的异常。最后利用Alphafold2数据库[16]中MINAR2蛋白结构预测的结果,并与胆固醇开展分子对接,研究发现MINAR2与胆固醇分子可以通过特定的氨基酸位点互作,且这些氨基酸位点集中在分析比对得到的CSD保守区段(图4)。综上提示MINAR2通过特定的氨基酸序列结合胆固醇。

图4 MINAR2结构模型与胆固醇之间的分子对接

图4 MINAR2结构模型与胆固醇之间的分子对接
至此,研究人员得到了如下的工作模型(图5):Minar2通过结合胆固醇,并参与胆固醇向纤毛束的转输,从而调节胆固醇在毛细胞中的稳态,进而保证毛细胞的正常发育。当minar2基因缺失后,胆固醇不能被运输至纤毛束,进而导致毛细胞纤毛束异常,伴随毛细胞顶端溶酶体异常,以及毛细胞的死亡。
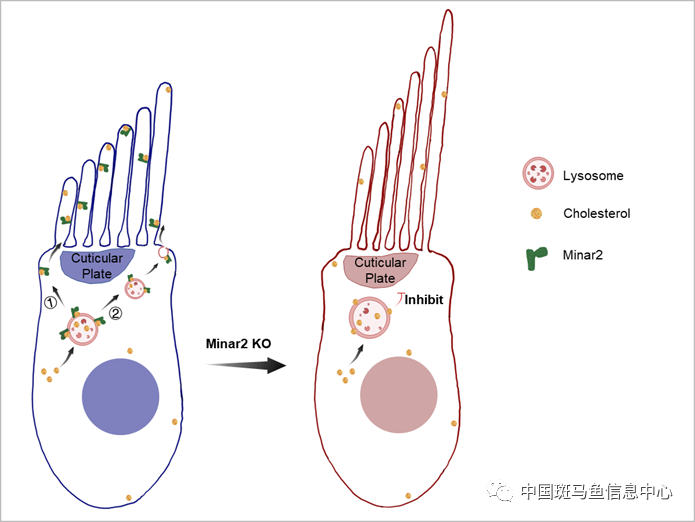
图5 工作模型

图5 工作模型
综上所述,本论文发现胆固醇在听觉毛细胞的纤毛束中富集并发挥关键作用,而脊椎动物所特有的Minar2基因通过调控胆固醇的运输及稳态从而保证正常的听力。论文结果还提示通过干预毛细胞中胆固醇有望恢复人类中携带MINAR2基因突变耳聋病人的听力。此外,近期的一项筛选研究发现Minar2基因家族另一成员KIAA1024/MINAR1/UBTOR在新冠病毒SARS-Co-V2的复制中是必需的宿主因子[17]。结合课题组之前对KIAA1024/MINAR1/UBTOR的研究[18],本论文结果提示胆固醇分子、以及在细胞内特定亚细胞器有可能成为抑制SARS-Co-V2在胞内复制的新靶点。
论文第一作者为复旦大学脑科学研究院博士研究生高鸽;通讯作者是复旦大学脑科学研究院彭刚研究员。该研究得到了国家重点研发计划和国家自然科学基金等项目的支持。
参考文献
1. Brown, C. S., Emmett, S. D., Robler, S. K., & Tucci, D. L. (2018). Global Hearing Loss Prevention. Otolaryngologic clinics of North America, 51(3), 575–592.
2. Fettiplace, R. (2017). Hair Cell Transduction, Tuning, and Synaptic Transmission in the Mammalian Cochlea. Compr Physiol, 7(4), 1197-1227.
3. Hudspeth, A. J. (1989). How the ear's works work. Nature, 341(6241), 397-404.
4. O. Maoileidigh, D., & Ricci, A. J. (2019). A Bundle of Mechanisms: Inner-Ear Hair-Cell Mechanotransduction. Trends Neurosci, 42(3), 221-236.
5. Bowl, M. R., Simon, M. M., Ingham, N. J., Greenaway, S., Santos, L., Cater, H., Taylor, S., Mason, J., Kurbatova, N., Pearson, S., Bower, L. R., Clary, D. A., Meziane, H., Reilly, P., Minowa, O., Kelsey, L., International Mouse Phenotyping, C., Tocchini-Valentini, G. P., Gao, X., . . . Brown, S. D. M. (2017). A large scale hearing loss screen reveals an extensive unexplored genetic landscape for auditory dysfunction. Nat Commun, 8(1), 886.
6. Ingham, N. J., Pearson, S. A., Vancollie, V. E., Rook, V., Lewis, M. A., Chen, J., Buniello, A., Martelletti, E., Preite, L., Lam, C. C., Weiss, F. D., Powis, Z., Suwannarat, P., Lelliott, C. J., Dawson, S. J., White, J. K., & Steel, K. P. (2019). Mouse screen reveals multiple new genes underlying mouse and human hearing loss. PLoS Biol, 17(4), e3000194.
7. Bademci, G., Lachgar-Ruiz, M., Deokar, M., Zafeer, M. F., Abad, C., Yildirim Baylan, M., Ingham, N. J., Chen, J., Sineni, C. J., Vadgama, N., Karakikes, I., Guo, S., Duman, D., Singh, N., Harlalka, G., Jain, S. P., Chioza, B. A., Walz, K., Steel, K. P., Nasir, J., … Tekin, M. (2022). Mutations in MINAR2 encoding membrane integral NOTCH2-associated receptor 2 cause deafness in humans and mice. Proceedings of the National Academy of Sciences of the United States of America, 119(26), e2204084119.
8. Revelo, N. H., Kamin, D., Truckenbrodt, S., Wong, A. B., Reuter-Jessen, K., Reisinger, E., Moser, T., & Rizzoli, S. O. (2014). A new probe for super-resolution imaging of membranes elucidates trafficking pathways. J Cell Biol, 205(4), 591-606.
9. Spicer, S. S., Thomopoulos, G. N., & Schulte, B. A. (1999). Novel membranous 1279 structures in apical and basal compartments of inner hair cells. J Comp Neurol, 1280 409(3), 424-437.
10. Wiwatpanit, T., Remis, N. N., Ahmad, A., Zhou, Y., Clancy, J. C., Cheatham, M. A., & Garcia-Anoveros, J. (2018). Codeficiency of Lysosomal Mucolipins 3 and 1 in Cochlear Hair Cells Diminishes Outer Hair Cell Longevity and Accelerates Age-Related Hearing Loss. J Neurosci, 38(13), 3177-3189.
11. Das, A., Brown, M. S., Anderson, D. D., Goldstein, J. L., & Radhakrishnan, A. (2014). Three pools of plasma membrane cholesterol and their relation to cholesterol homeostasis. Elife, 3.
12. Lim, C. Y., Davis, O. B., Shin, H. R., Zhang, J., Berdan, C. A., Jiang, X., Counihan, J. L., Ory, D. S., Nomura, D. K., & Zoncu, R. (2019). ER-lysosome contacts enable cholesterol sensing by mTORC1 and drive aberrant growth signalling in Niemann-Pick type C. Nat Cell Biol, 21(10), 1206-1218.
13. Maekawa, M., & Fairn, G. D. (2015). Complementary probes reveal that phosphatidylserine is required for the proper transbilayer distribution of cholesterol. J Cell Sci, 128(7), 1422-1433.
14. Naito, T., Ercan, B., Krshnan, L., Triebl, A., Koh, D. H. Z., Wei, F. Y., Tomizawa, K., Torta, F. T., Wenk, M. R., & Saheki, Y. (2019). Movement of accessible plasma membrane cholesterol by the GRAMD1 lipid transfer protein complex. Elife, 8.
15. Schoop, V., Martello, A., Eden, E. R., & Hoglinger, D. (2021). Cellular cholesterol and how to find it. Biochim Biophys Acta Mol Cell Biol Lipids, 1866(9), 158989.
16. Tunyasuvunakool, K., Adler, J., Wu, Z., Green, T., Zielinski, M., ?ídek, A., Bridgland, A., Cowie, A., Meyer, C., Laydon, A., Velankar, S., Kleywegt, G. J., Bateman, A., Evans, R., Pritzel, A., Figurnov, M., Ronneberger, O., Bates, R., Kohl, S. A. A., . . . Hassabis, D. (2021). Highly accurate protein structure prediction for the human proteome. Nature, 596(7873), 590-596.
17. Kratzel, A., Kelly, J. N., V'Kovski, P., Portmann, J., Bruggemann, Y., Todt, D., Ebert, N., Shrestha, N., Plattet, P., Staab-Weijnitz, C. A., von Brunn, A., Steinmann, E., Dijkman, R., Zimmer, G., Pfaender, S., & Thiel, V. (2021). A genome-wide CRISPR screen identifies interactors of the autophagy pathway as conserved coronavirus targets. PLoS Biol, 19(12), e3001490.
18. Zhang, H., Zhang, Q., Gao, G., Wang, X., Wang, T., Kong, Z., Wang, G., Zhang, C., Wang, Y., & Peng, G. (2018). UBTOR/KIAA1024 regulates neurite outgrowth and neoplasia through mTOR signaling. PLoS Genet, 14(8), e1007583.