孟安明组揭示核孔复合体调控合子基因组激活新机制
作者:CZRC 发布时间:2022/12/9 11:00:00
核孔复合体(nuclear pore complex, NPC)是生物体内已知的最大复合体,一般认为NPC是真核细胞内细胞核与细胞质之间生物大分子的唯一运输通道,其结构在不同物种中高度相似,均为八倍辐射对称结构。作为结构生物学领域的“圣杯”之一,核孔复合体的真容正变得更加清晰(Science | 揭开“圣杯”面纱:Hoelz团队两文解析核孔复合物多个精细构象;Science丨揭开“圣杯”面纱:施一公团队揭示核孔复合物胞质环的高分辨率冷冻电镜结构;Cell | 酵母核孔复合物的综合结构和功能适应;里程碑丨施一公团队解析核孔复合物近原子分辨率胞质环结构及高分辨率原位核孔复合物腔环结构),例如,同一物种不同类型的细胞中,核孔的组成存在差异[1];同一细胞中,核孔的组成与结构也不完全一致[2];并且组成成分相同的核孔复合体,外界环境会影响其构象,从而改变其核-质转运的能力[3]。目前已知核孔复合体对于细胞的命运决定具有重要意义[4];且已报道多种核孔蛋白基因突变后,可引起动物组织特异性缺陷或致死[5]。但是,目前尚不清楚受精后早期胚胎发育过程中,核孔复合体的组成与构象是否发生改变,以及其是否调控了重要的生物学过程。
2022年12月8日,清华大学生命科学学院孟安明院士课题组于Cell在线发表论文"Comprehensive maturity of nuclear pore complexes regulates zygotic genome activation",报道了核孔复合体综合成熟度调控合子基因组激活的全新机制。


胚胎发育和细胞分化均依赖于高度协调的基因表达调控。几乎所有的后生动物,在受精后的数小时内,早期胚胎发育完全依赖于卵子来源的RNA和蛋白,而合子基因组保持沉默无转录发生。一段时间后,大量的合子基因同时开始表达,称为合子基因组激活(zygotic genome activation, 简称major ZGA);在这之前,会有少量的合子基因表达,称为minor ZGA;同时母源产物也开始逐渐降解;整个过程称为母源合子转换(maternal to zygotic transition, 简称MZT)。MZT过程中,高度分化的雌雄配子重编程为具有多能性甚至全能性的胚胎干细胞。早在上世纪60年代,John B. Gurdon将高度分化的肠细胞细胞核移植入去核的蛙卵细胞,成功获得了蝌蚪[6],证明了母源物质对细胞多能性的重要作用。但是,核移植的成功率并不高,这可能与基因组重编程相关。2006年,Shinya Yamanaka通过转基因过表达四个转录因子,成功将高度分化的细胞诱导为多能性干细胞[8]。John B. Gurdon和Shinya Yamanaka因其重要工作,共同获得了2012年诺贝尔生理学与医学奖。因此,了解MZT过程中合子基因组激活的机制,有利于理解细胞多能性建立的分子机理,为干细胞研究和再生医学等的发展提供理论指导。
ZGA的发生时间在不同物种中存在差异,但是在同一物种中高度保守,如果蝇、爪蟾和斑马鱼等物种发生在囊胚中期,在此之前胚盘细胞基本同步快速分裂,然后大量合子基因开始转录表达,细胞分裂变得不同步,该时期也称为中囊胚转换期(midblastula transition, 简称MBT);小鼠发生在2细胞期;而人发生在8细胞期。该研究所用的斑马鱼,minor ZGA开始于128细胞期(128c, 2.25 hour postfertilization [hpf]),major ZGA/MBT则大约开始于受精后第10次卵裂,也就是512细胞期(512c, 2.75 hpf)至1k细胞期(1kc, 3 hpf)。目前已经鉴定确认母源转录因子Nanog、Pou5f1和Sox19b对于斑马鱼minor ZGA少量合子基因的转录及MBT时期大量合子基因组激活均具有重要作用[8,9]。其它卵子来源的转录因子如Eomesodermin a (Eomesa)等也要到MBT时期才能激活下游合子基因的表达[10]。那么不同的转录因子,为什么都要到MBT才能激活下游合子基因的表达?染色质结构、母源蛋白修饰或缺乏必要的辅助因子等因素有可能调控合子基因转录,但是这些因素很难解释大量合子基因在同一时间点开始转录这一现象。核孔复合体作为生物大分子的核-质转运的唯一通道,有可能通过调控母源转录因子的入核而调控了ZGA的发生。该研究以斑马鱼为模式动物,发现从受精后至MBT的早期胚胎中,核孔复合体的组成、大小和转运能力等均逐渐增加,即核孔复合体综合成熟度(comprehensive NPC maturity, 简称CNM)逐渐增加,其介导母源转录因子的入核量逐渐升高,从而调控ZGA的发生。CNM有可能是ZGA发生的分子时钟。
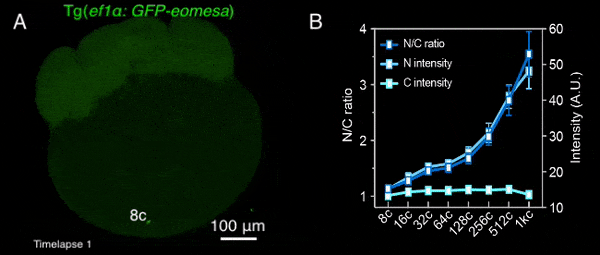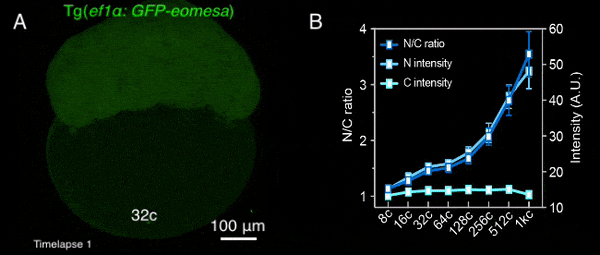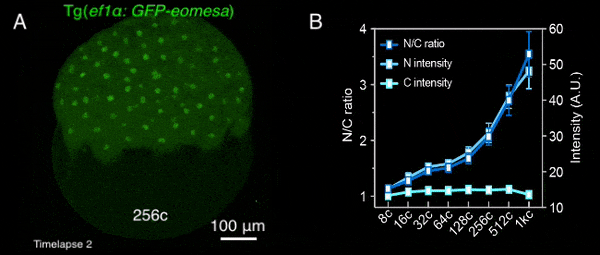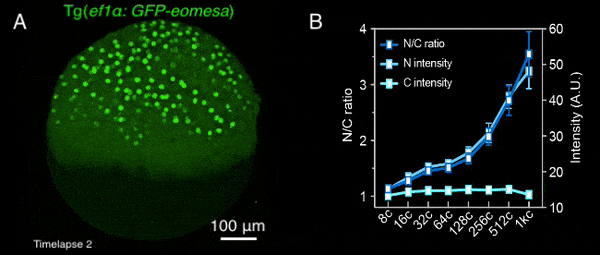
图1 转基因母源过表达的Eomesa在早期胚胎中入核逐渐增加

图1 转基因母源过表达的Eomesa在早期胚胎中入核逐渐增加
该研究首先通过定量质谱分析了MBT前不同发育时期,转录因子和核孔蛋白的细胞核定位情况,发现包括Nanog、Pou5f1、Sox19b和Eomesa在内的大部分母源转录因子,其在细胞核中的量随胚胎发育显著增加,预示着其可能通过剂量依赖模式调控了ZGA的发生。核孔蛋白也表现出类似的分布特征,总的来说,随着早期胚胎发育,更多的核孔蛋白存在于细胞核,即可能组装形成核孔复合体。后续一系列实验进一步验证了该结果,包括母源转录因子Eomesa、Nanog、pSmad1/5/9等的入核量均在MBT前随着胚胎发育逐渐增加(图1),体外合成携带核定位信号的荧光蛋白也表现出了类似的入核变化趋势,并且在MBT后达到峰值且基本保持不变。这种随早期胚胎发育改变的入核趋势,与出核转运、被动扩散或蛋白翻译积累的关系不大,而核孔复合体的入核转运功能在这一早期发育过程中逐渐增强。同时,在受精后至MBT这一阶段,核孔复合体的组成逐渐变得更加复杂,尺寸也总体上增大,成熟核孔的比例逐渐增加,即CNM逐渐增加,在MBT后进入最大值,其后基本保持不变(图2)。有趣的是,CNM的这种变化趋势与母源转录因子或核定位信号介导的荧光蛋白的入核变化趋势高度一致,且CNM的变化曲线要早于核蛋白的入核量变化,预示着CNM可能是斑马鱼早期胚胎发育过程中母源转录因子入核变化的关键调控因素之一。
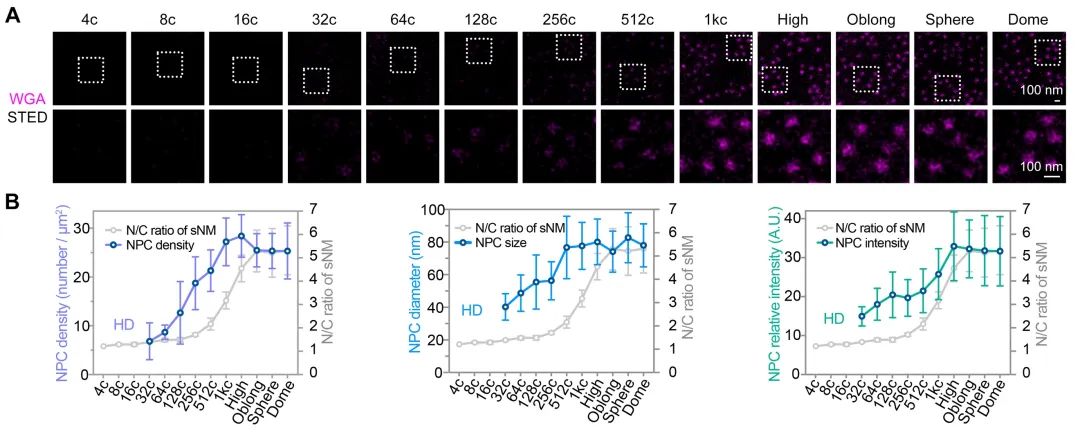
图2 核孔复合体综合成熟度随早期胚胎发育逐渐增加

图2 核孔复合体综合成熟度随早期胚胎发育逐渐增加
接下来,该研究通过基因敲除核孔蛋白基因nup133和elys,或转基因过表达nup133,成功干扰或促进了核孔复合体在早期胚胎中的组装。在突变体胚胎中,母源转录因子的入核被显著干扰,合子基因的表达明显延迟;而转基因过表达则明显促进了转录因子的入核与合子基因的转录激活。综上所述,在斑马鱼早期胚胎发育过程中,核孔复合体综合成熟度逐渐增加,通过介导母源转录因子的入核变化,其调控了合子基因组激活的发生,扮演了ZGA分子时钟的角色(图3)。
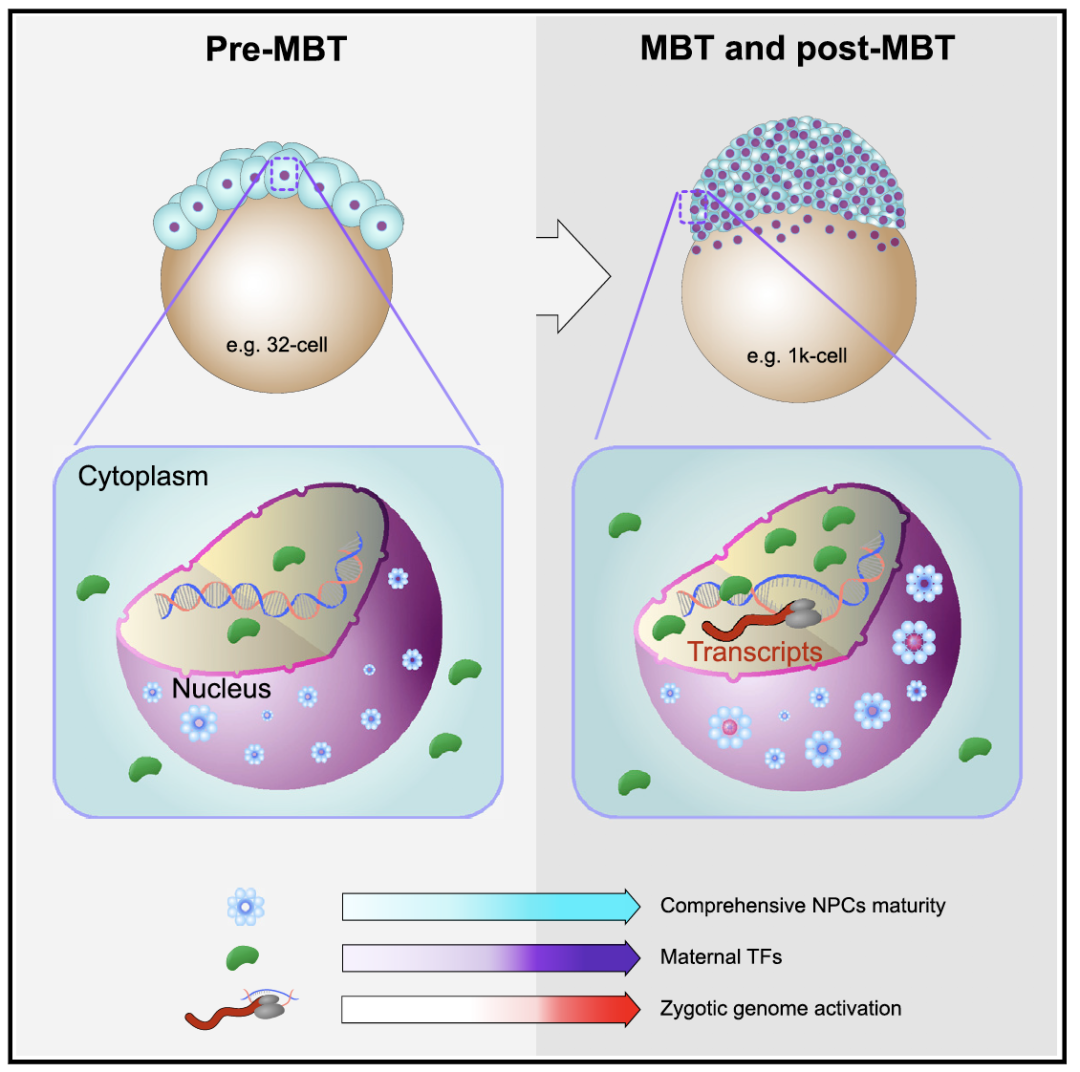
图3 核孔复合体综合成熟度调控合子基因组激活模式图

图3 核孔复合体综合成熟度调控合子基因组激活模式图
关于ZGA发生的机制,以前主要有两种模型:核质比模型和母源时钟模型。该研究所提出的CNM可以认为是对母源时钟模型的重要补充,同时CNM也可能受到核质比模型与母源时钟模型的调控。该课题无论对核孔复合体的功能研究,还是对合子基因组激活的机制解释都提供了新的视角,阐明了许多重要的生物学问题,同时也提供了许多新的研究兴趣。比如,核孔复合体综合成熟度变化的调控因素是什么?核孔复合体的分子社会学变化是怎样的?ZGA中是否存在不依赖核孔复合体成熟度的转录因子?这一现象在其它物种中是否保守存在?回答这些问题将有利于我们更深入理解生物大分子复合物参与重要生物学事件的过程与机理。
清华大学生命学院孟安明院士为论文通讯作者,课题组博士后沈炜敏为第一作者。
参考文献
1. Raices, M., and D'Angelo, M.A. (2012). Nuclear pore complex composition: a new regulator of tissue-specific and developmental functions. Nature Reviews Molecular Cell Biology 13, 687. 10.1038/nrm3461.
2. Akey, C.W., Singh, D., Ouch, C., Echeverria, I., Nudelman, I., Varberg, J.M., Yu, Z., Fang, F., Shi, Y., Wang, J., et al. (2022). Comprehensive structure and functional adaptations of the yeast nuclear pore complex. Cell 185, 361-378.e325. https://doi.org/10.1016/j.cell.2021.12.015.
3. Zimmerli, C.E., Allegretti, M., Rantos, V., Goetz, S.K., Obarska-Kosinska, A., Zagoriy, I., Halavatyi, A., Hummer, G., Mahamid, J., Kosinski, J., and Beck, M. (2021). Nuclear pores dilate and constrict in cellulo. Science 0, eabd9776. doi:10.1126/science.abd9776.
4. Gomar-Alba, M., and Mendoza, M. (2020). Modulation of cell identity by modification of nuclear pore complexes. Frontiers in Genetics 10, 1301.
5. Guglielmi, V., Sakuma, S., and D'Angelo, M.A. (2020). Nuclear pore complexes in development and tissue homeostasis. Development 147, dev183442. 10.1242/dev.183442.
6. Gurdon, J.B. (1962). The Developmental Capacity of Nuclei taken from Intestinal Epithelium Cells of Feeding Tadpoles. Development 10, 622-640. 10.1242/dev.10.4.622.
7. Takahashi, K., and Yamanaka, S. (2006). Induction of Pluripotent Stem Cells from Mouse Embryonic and Adult Fibroblast Cultures by Defined Factors. Cell 126, 663-676. 10.1016/j.cell.2006.07.024.
8. Lee, M.T., Bonneau, A.R., Takacs, C.M., Bazzini, A.A., DiVito, K.R., Fleming, E.S., and Giraldez, A.J. (2013). Nanog, Pou5f1 and SoxB1 activate zygotic gene expression during the maternal-to-zygotic transition. Nature 503, 360-364. 10.1038/nature12632.
9. Leichsenring, M., Maes, J., M?ssner, R., Driever, W., and Onichtchouk, D. (2013).Pou5f1 Transcription Factor Controls Zygotic Gene Activation In Vertebrates. Science 341, 1005-1009. 10.1126/science.1242527.
10. Xu, P., Zhu, G., Wang, Y., Sun, J., Liu, X., Chen, Y.-G., and Meng, A. (2014). Maternal Eomesodermin regulates zygotic nodal gene expression for mesendoderm induction in zebrafish embryos. J. Mol. Cell. Biol. 6, 272-285. 10.1093/jmcb/mju028.
参考文献
1. Raices, M., and D'Angelo, M.A. (2012). Nuclear pore complex composition: a new regulator of tissue-specific and developmental functions. Nature Reviews Molecular Cell Biology 13, 687. 10.1038/nrm3461.
2. Akey, C.W., Singh, D., Ouch, C., Echeverria, I., Nudelman, I., Varberg, J.M., Yu, Z., Fang, F., Shi, Y., Wang, J., et al. (2022). Comprehensive structure and functional adaptations of the yeast nuclear pore complex. Cell 185, 361-378.e325. https://doi.org/10.1016/j.cell.2021.12.015.
3. Zimmerli, C.E., Allegretti, M., Rantos, V., Goetz, S.K., Obarska-Kosinska, A., Zagoriy, I., Halavatyi, A., Hummer, G., Mahamid, J., Kosinski, J., and Beck, M. (2021). Nuclear pores dilate and constrict in cellulo. Science 0, eabd9776. doi:10.1126/science.abd9776.
4. Gomar-Alba, M., and Mendoza, M. (2020). Modulation of cell identity by modification of nuclear pore complexes. Frontiers in Genetics 10, 1301.
5. Guglielmi, V., Sakuma, S., and D'Angelo, M.A. (2020). Nuclear pore complexes in development and tissue homeostasis. Development 147, dev183442. 10.1242/dev.183442.
6. Gurdon, J.B. (1962). The Developmental Capacity of Nuclei taken from Intestinal Epithelium Cells of Feeding Tadpoles. Development 10, 622-640. 10.1242/dev.10.4.622.
7. Takahashi, K., and Yamanaka, S. (2006). Induction of Pluripotent Stem Cells from Mouse Embryonic and Adult Fibroblast Cultures by Defined Factors. Cell 126, 663-676. 10.1016/j.cell.2006.07.024.
8. Lee, M.T., Bonneau, A.R., Takacs, C.M., Bazzini, A.A., DiVito, K.R., Fleming, E.S., and Giraldez, A.J. (2013). Nanog, Pou5f1 and SoxB1 activate zygotic gene expression during the maternal-to-zygotic transition. Nature 503, 360-364. 10.1038/nature12632.
9. Leichsenring, M., Maes, J., M?ssner, R., Driever, W., and Onichtchouk, D. (2013).Pou5f1 Transcription Factor Controls Zygotic Gene Activation In Vertebrates. Science 341, 1005-1009. 10.1126/science.1242527.
10. Xu, P., Zhu, G., Wang, Y., Sun, J., Liu, X., Chen, Y.-G., and Meng, A. (2014). Maternal Eomesodermin regulates zygotic nodal gene expression for mesendoderm induction in zebrafish embryos. J. Mol. Cell. Biol. 6, 272-285. 10.1093/jmcb/mju028.